Anotación del genoma de Microtom y comparación con Heinz y otras especies. Crédito: Investigación en horticultura
El tomate (Solanum lycopersicum) ha reconocido su importancia mundial con una producción anual estimada de 186 millones de toneladas en 2020. Además de su valor comestible y económico, el tomate es un modelo importante en la investigación de la biología del desarrollo, superando a Arabidopsis thaliana en algunos contextos como el desarrollo del fruto. , metabolismo, interacciones planta-patógeno y estudios simbióticos.
Microtom es un cultivar de tomate y actualmente una planta modelo experimental ampliamente aplicada para estudios de laboratorio debido a su pequeño tamaño, ciclo de crecimiento corto y alta eficiencia de transformación.
A pesar de la extensa resecuenciación de varios cultivares de tomate, un genoma de alta calidad para Microtome está notoriamente ausente, lo que presenta un desafío para los biólogos del desarrollo que dependen del genoma del tomate Heinz como referencia pero usan Microtome para experimentos funcionales.
En julio de 2023, Investigación en horticultura Microtom publicó un artículo de investigación titulado «Redes reguladoras integradas para el desarrollo de órganos del tomate basadas en el genoma y el ARNoma del tomate». En este estudio, los investigadores proporcionaron un genoma de Microtome de alta calidad y realizaron un análisis genómico comparativo con el genoma del tomate Heinz publicado anteriormente.
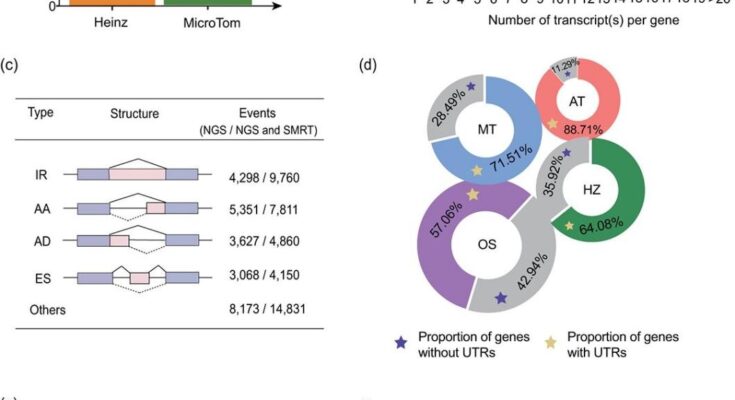
Primero, obtuvieron un ensamblaje del genoma de un microtomo de 799 Mb con 60 cóntigos utilizando una combinación de datos de secuenciación de Nanopore e Illumina. La anotación del gen codificador de proteínas del ensamblaje del microtomo captura el 98,57% de los genes de Embryophyta BUSCO (odb10).
La genómica comparada reveló similitudes y diferencias sustanciales entre los genomas del Microtomo y de Heinz, ambos comparten eventos de poliploidía comunes pero exhiben divergencia genómica sustancial en términos de contenido genético, diferencias estructurales y polimorfismos de un solo nucleótido (SNP).
Además, los investigadores presentan el panorama del ARNoma del microtomo en diferentes órganos/etapas de desarrollo/tratamientos y realizaron un análisis exhaustivo del transcriptoma de los genes codificadores de proteínas del microtomo en relación con la expresión génica y el empalme alternativo (AS).
Luego, construyeron múltiples redes de coexpresión genética con el genoma de referencia y abundantes datos de expresión genética, lo que proporcionó pistas valiosas para la identificación de genes clave implicados en diversas vías reguladoras durante el desarrollo de las plantas, como la simbiosis de micorrizas arbusculares y el desarrollo de los frutos.
El estudio arroja luz sobre los ARN no codificantes, incluidos los miARN, los lncRNA y los circRNA, al combinar datos de diferentes etapas y condiciones de desarrollo.
Se identificaron un total de 210 miARN a partir de los conjuntos de datos de sARN, de los cuales 164 pertenecían a 48 familias de miARN de tomate conocidas en miRbase. Un total de 4.835 lncRNA fueron anotados por transcriptomas de microtom y> 2.944 (60,89%) se transcribieron de regiones intergénicas. y se identificaron 19.840 circRNA respaldados por tres herramientas de software independientes mediante el mapeo de lecturas de secuencia en el genoma del microtom.
Finalmente, los investigadores desarrollaron una base de datos integral (Microdatabase) que brinda posibilidades de búsqueda y descarga en línea de sus datos y resultados. Este recurso en línea está diseñado para ser una herramienta invaluable para los investigadores que trabajan con tomates micrótomos, mejorando los estudios en genómica comparada, expresión genética y genómica funcional.
En resumen, este estudio presenta no solo un genoma de microtomía de alta calidad, sino también una rica descripción de elementos codificantes y no codificantes, respaldada por extensos análisis del transcriptoma.
Los conocimientos sobre los perfiles de expresión genética, el empalme alternativo, las funciones del ARN no codificante y las redes de coexpresión aumentarán significativamente nuestra comprensión de la complejidad genómica y transcriptómica del tomate microtom, proporcionando un recurso valioso para futuras investigaciones en biología y genética vegetal.
Más información:
Jia-Yu Xue et al, Redes reguladoras integradas para el desarrollo de órganos del tomate basadas en el genoma y el ARNoma del tomate en microtom, Investigación en horticultura (2023) DOI: 10.1093/h/uhad147
Proporcionado por Fenómica Vegetal
referencia: Unveiling a high-quality microtom genome and its impact on development biology (2024, 24 de enero) Consultado el 24 de febrero de 2024 en https://phys.org/news/2024-01-unveiling-high-quality-microtom-genome.html
Este documento está sujeto a derechos de autor. Ninguna parte puede reproducirse sin permiso por escrito, excepto en cualquier trato justo con fines de estudio o investigación privados. El contenido se proporciona únicamente con fines informativos.